Diversidad bacteriana presente en leche de cabras con mastitis clínica en Ecuador
Contenido principal del artículo
Resumen
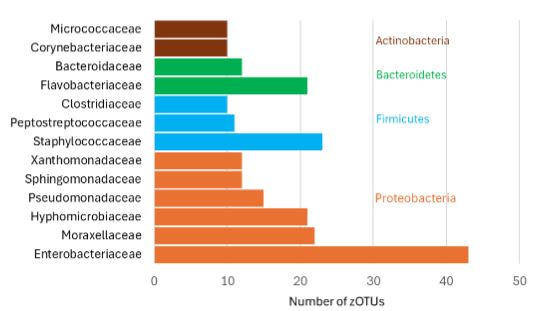
La mastitis es una inflamación de la glándula mamaria que afecta significativamente a la producción y calidad de la leche en las cabras, lo que genera pérdidas económicas para los agricultores ecuatorianos. Este estudio tuvo como objetivo explorar la diversidad bacteriana asociada con la mastitis clínica en cabras en Ecuador. Se recolectaron muestras de leche de cabras con evidencia macroscópica de mastitis clínica para posteriormente ser extraído el ADN. Para el análisis metagenómico se amplificó y secuenció el gen 16S rRNA mediante la tecnología Illumina MiSeq. Las secuencias se filtraron por calidad y agrupadas en unidades taxonómicas operacionales de radio cero (zOTUs); luego se agruparon taxonómicamente para clasificar las especies bacterianas presentes en la leche. El análisis reveló una alta riqueza de comunidades bacterianas, con 550 zOTUs pertenecientes a 12 filos. Proteobacteria y Firmicutes fueron los filos predominantes, los cuales incluyen diversas familias, como: Enterobacteriaceae (Proteobacteria) y Staphylococcaceae (Firmicutes). Notablemente, una porción significativa (86,91%) de los zOTUs identificados pertenecían a familias con funcionalidades desconocidas en el contexto de la mastitis. Estos hallazgos ofrecen una visión inicial y valiosa sobre la diversidad bacteriana asociada con la mastitis clínica en cabras, resaltando además la posible presencia de bacterias aún no caracterizadas que podrían ser relevantes en el contexto de esta enfermedad. Es necesario realizar futuros estudios que se enfoquen en la identificación a nivel de especie y la caracterización funcional, para desarrollar estrategias dirigidas a la prevención y control de la mastitis en la producción caprina ecuatoriana.
Detalles del artículo

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Aquellos autores/as que tengan publicaciones con esta revista, aceptan los términos siguientes:
- Los autores/as conservarán sus derechos de autor y garantizarán a la revista el derecho de primera publicación de su obra, el cual estará simultáneamente sujeto a la Licencia Creative Commons Attribution-NonCommercial 4.0 International (CC BY-NC 4.0) que prohíbe el uso comercial de sus publicaciones y permite a terceros compartir la obra siempre que se indique su autor y la primera publicación en esta revista. Bajo esta licencia el autor será libre de:
- Compartir — copiar y redistribuir el material en cualquier medio o formato
- Adaptar — remezclar, transformar y crear a partir del material
- El licenciador no puede revocar estas libertades mientras cumpla con los términos de la licencia
Bajo las siguientes condiciones:
- Reconocimiento — Debe reconocer adecuadamente la autoría, proporcionar un enlace a la licencia e indicar si se han realizado cambios. Puede hacerlo de cualquier manera razonable, pero no de una manera que sugiera que tiene el apoyo del licenciador o lo recibe por el uso que hace.
- NoComercial — No puede utilizar el material para una finalidad comercial.
- No hay restricciones adicionales — No puede aplicar términos legales o medidas tecnológicas que legalmente restrinjan realizar aquello que la licencia permite.
- Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
- Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos telemáticos institucionales o en su página web) antes y durante el proceso de envío, lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).
Citas
FAO. (2020) Dairy Market Review - Overview of Global Dairy Market Developments in 2019.
Prosser CG. Navigating Food Analysis: Practical Tips and Tricks from Sample Prep to LC. J. Food Sci. 2021; 86: 257-265. https:// doi.org/10.1111/1750-3841.15574].
ALKaisy QH, Al-Saadi J, Al-Rikabi AK, Altemimi AB, Hesarinejad A, Abeledmaksoud TG. Exploring the health benefits and functional properties of goat milk proteins. Food Sci Nutr. 2023; 11(10): 5641-5656. doi: 10.1002/fsn3.3531.
Mirzaei H, Chaleshtori RS. Role of fermented goat milk as a nutritional product to improve anemia, J Food Biochem. 2021; 46, e13969. https://doi.org/ 10.1111/jfbc.13969
Ajmone-Marsan P, Colli L, Han JL, Achilli A, Lancioni H, Joost S, Crepaldi P, Pilla F, Stella A, Taberlet P, Boettcher P, Negrini R, Lenstra JA. The characterization of goat genetic diversity: Towards a genomic approach. Small Rumin Res. 2014; 121(1):58-72. doi:10.1016/j.smallrumres.2014.06.010.
Ginja C, Cortés O, Gama LT, Delgado JV, Amills M, De Sousa C, et al. (2018) Conservation of Goat Populations from Southwestern Europe Based on Molecular Diversity Criteria. In: Simões J, Gutiérrez C, eds. Sustainable Goat Production in Adverse Environments: Volume I. Springer, Cham pp 509-5335. doi:10.1038/ismej.2011.41.
Nguyen VD, Nguyen CO, Chau TML, Nguyen DQD, Han AT, Le TTH. Goat Production, Supply Chains, Challenges, and Opportunities for Development in Vietnam: A Review. Animals. 2023;13(15):2546. doi: 10.3390/ani13152546
Polveiro RC, Vidigal PM, Mendes TA, Yamatogi RS, Lima MC, Moreira MA. Effects of enrofloxacin treatment on the bacterial microbiota of milk from goats with persistent mastitis. Sci Rep. 2020; 10(1):4421. doi:10.1038/s41598-020-61407-2.
Oikonomou G, Bicalho ML, Meira E, Rossi RE, Foditch C, Machado VS, Teixeira AG, Santisteban C, Schukken YH, Bicalho RC. Microbiota of Cow’s Milk; Distinguishing Healthy, Sub-Clinically and Clinically Diseased Quarters. Guan LL, ed. PLoS ONE. 2014; 9(1): e85904. doi:10.1371/journal.pone.0085904.
Azooz MF, El-Wakeel SA, Yousef HM. Financial and economic analyses of the impact of cattle mastitis on the profitability of Egyptian dairy farms. Vet World. 2020; 13(9):1750-1759. doi: 10.14202/vetworld.2020.1750-1759.
Podhorecká K, Borková M, Šulc M, Šulc M, Seydlová R, Hedvika Dragounová, H, Švejcarová M, Peroutková J, Elich O. Somatic Cell Count in Goat Milk: An Indirect Quality Indicator. Foods. 2021; 10(5), 1046; https://doi.org/10.3390/foods10051046.
Menzies P. Udder Health for Dairy Goats. Veterinary Clinics: Food Animal Practice. 2021; 37:1, 149 - 174
Novac CS, Andrei S. The Impact of Mastitis on the Biochemical Parameters, Oxidative and Nitrosative Stress Markers in Goat’s Milk: A Review. Pathogens. 2020; 9(11):882. doi:10.3390/pathogens9110882.
Rahimi H, Tukmechi A, Rashidian E. Genetic diversity of Brucella melitensis isolates from sheep and goat milk in Iran. Veterinary Research Forum. 2023; 14 (12): 649-657 doi: 10.30466/vrf.2023.1988859.3768
Contreras A, Sierra D, Sánchez A, Corrales JC, Marco JC, Paape MJ, Gonzalo C. Mastitis in small ruminants. Small Rumin Res. 2007; 68(1-2):145-153. doi:10.1016/j.smallrumres.2006.09.011.
Polveiro RC, Vidigal PMP, De Oliveira Mendes TA, Yamatogi RS, Da SilvaLS, Fujikura JM, et al. Distinguishing the milk microbiota of healthy goats and goats diagnosed with subclinical mastitis, clinical mastitis, and gangrenous mastitis. Front Microbiol. 2022; 13:918706. doi:10.3389/fmicb.2022.918706.
Deddefo A, Mamo G, Leta S, Amenu K. Prevalence and molecular characteristics of Sthaphylococcus aureus in raw milk and milk products in Ethiopia: a systematic review and meta-analisis. Int. J. Food Contam. 2022; 9:8 https://doi.org/ 10.1186/s40550-022-00094-5
Nagao PE, BurkovskiA, Mattos-Guaraldi AL. Editorial: Streptococcus spp. and Corynebacterium spp.: Clinical and Zoonotic Epidemiology, Virulence Potential, Antimicrobial Resistance, and Genomic Trends and Approaches. Front Microbiol. 2022; 28 (13):867210. doi: 10.3389/fmicb.2022.867210
McInnisEA, Kalanetra KM, Mills DA, Maga EA. Analysis of raw goat milk microbiota: Impact of stage of lactation and lysozyme on microbial diversity. Food Microbiol. 2015; 46:121-131. doi:10.1016/j.fm.2014.07.021.
Zhang F, Wang Z, Lei F, Wang Z, Jiang S, Peng Q, Zhang J, Shao Y. Bacterial diversity in goat milk from the Guanzhong area of China. J Dairy Sci. 2017; 100 (10):7812-7824. doi:10.3168/jds.2017-13244.
Ngara TR, Zhang H. Recent Advances in Function-Based Metagenomic Screening. Genomics Proteomics Bioinformatics. 2018; 16 (6):405-415. doi:10.1016/j.gpb.2018.01.002.
Ahmad T, Singh RS, Gupta G, Sharma A, Kaur B. Metagenomics in the Search for Industrial Enzymes. In: Advances in Enzyme Technology. Elsevier. 2019; 419-451.
Wright RJ, Gibson MI, Christie-Oleza JA. Understanding microbial community dynamics to improve optimal microbiome selection. Microbiome. 2019; 7(1):85. doi:10.1186/s40168-019-0702-x.
Win Epi (Working in epidemiology) 2024. [Citado Sept 29]. Disponible en: http:// www.winepi.net/sp/index.htm
ESPAC (Instituto Ecuatoriano de Estadísticas y Censos) 2023 [Citado Sept 29]. Disponible en: https://www.ecuadorencifras.gob.ec/estadisticasagropecuarias-2/
Herlemann, DP, Labrenz M, Jürgens K, Bertilsson S, Waniek JJ, Andersson AF. Transitions in bacterial communities along the 2000 km salinity gradient of the Baltic Sea. ISME J. 2011; 5(10):1571-1579. doi:10.1038/ismej.2011.41.
Andrés S. FastQC: A Quality Control tool for High Throughput Sequence Data. 2012. https:// www.bioinformatics.babraham.ac.uk/projects/fastqc/
Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014; 30 (15):2114-2120. doi:10.1093/bioinformatics/btu170.
Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nat Methods. 2013; 10(10):996-998.
Edgar RC. Updating the 97% identity threshold for 16S ribosomal RNA OTUs. Valencia A, ed. Bioinformatics. 2018; 34(14):2371-2375. doi:10.1038/nmeth.2604.
Herrera P, Suárez JP, Sánchez-Rodríguez, A, Molina MC, Prieto M, Méndez M. Many broadly-shared mycobionts characterize mycorrhizal interactions of two coexisting epiphytic orchids in a high elevation tropical forest. Fungal Ecol. 2019; 39:26-36. doi:10.1016/j.funeco.2018.11.003.
Edgar RC. UNOISE2: improved error-correction for Illumina 16S and ITS amplicon sequencing. BioRxiv, 2016. doi:10.1093/bioinformatics/bty113.
Callahan BJ, McMurdie PJ, Rosen MJ, Han AW, Johnson AJA, Holmes SP. DADA2: High-resolution sample inference from Illumina amplicon ta. Nat Methods. 2016; 13(7):581-583. doi:10.1038/nmeth.3869.
SoberónM J, Llorente BJ. The Use of Species Accumulation Functions for the Prediction of Species Richness. Conserv Biol.1993; 7(3):480-488. doi:10.1046/j.1523-1739.1993.07030480.x.
Jiménez-ValverdeA, Horta J Las curvas de acumulación de especies y la necesidad de evaluar la calidad de los inventarios biológicos. Rev Ibérica Aracnol2003; 8:151-161.
Anil,N.S, JanusA., Deepa, P.M. Shyma VH, RajasekharR., Habeeb BB. Anand LF. Clinicopathological findings and etiological characterization in caprine gangrenous mastitis. Small Rumin Res. 2024 241: 107389 https://doi.org/ 10.1016/j.smallrumres.2024.107389
RibeiroMG, Lra GHB, Bicudo SD, Souza AVG, Salerno T, Siqueira AK, GeraldoJS. An unusual gangrenous goat mastitis caused by Staphylococcus aureus, Clostridium perfringens and Escherichia coli co-infection. Arq Bras Med Veterinária E Zootec. 2007; 59(3):810-812. doi:10.1590/S0102-09352007000300037.
Niyazbekova Z, Yao XT, Liu MJ, Bold N, Tong JZ, Chang Jj, Wen Y, Li L, WangY, Chen DK, Ma WT. Compositional and functional comparison of the microbiota in the colostrum and mature Milk of dairy goats. Animals. 2020; 10(11): 1955 https://doi.org/ 10.3390/ani10111955
Quigley L, O’Sullivan O, Stanton C, Beresford TP, Ross RP, Fitzgerald GF, Cotter PD. The complex microbiota of raw milk. FEMS Microbiol Rev. 2013; 37(5):664-698. doi:10.1111/1574-6976.12030.
Breitenwieser F, Doll EV, Clavel T, Scherer S, Wenning M. Complementary Use of Cultivation and High-Throughput Amplicon Sequencing Reveals High Biodiversity Within Raw Milk Microbiota. Front Microbiol. 2020; 11:1557. doi:10.3389/fmicb.2020.01557.
Nelli A, Voidarou C, Venardou B, Fotou K, Tsinas A, Bonos E, Fthenakis GC, Skoufos I, Tzora A. Antimicrobial and Methicillin Resistance Pattern of Potential Mastitis-Inducing Staphylococcus aureus and Coagulase-Negative Staphylococci Isolates from the Mammary Secretion of Dairy Goats. Biology. 2022; 11(11):1591. doi:10.3390/biology11111591.
Jabbar A, Saleem MH, Iqbal MZ, Qasim M, Ashraf M, Tolba MM, et al. Epidemiology and antibiogram of common mastitis-causing bacteria in Beetal goats. Vet World. 2020; 13(12):2596-2607. doi:10.14202/vetworld.2020.2596-2607.
Pereira CS, Santos LM, Machado LS, Melo DA, Coelho S, Pereira V, Souza M, Nascimiento E. Proteomics characterization of Staphylococcus spp. from goat mastitis and phenogeno-typical assessment of resistance to beta-lactamics. Pesqui Veterinária Bras. 2021; 41:e06129. doi:10.1590/1678-5150pvb-6129.
Hoving-Bolink RA, Antonis AF, te Pas MFW, Schokker D. An observational study of the presence and variability of the microbiota composition of goat herd milk related to mainstream and artisanal farm management. PLOS ONE. 2023; 18(10): e0292650. https://doi.org/10.1371/journal.pone.0292650.
Tormo H, Delacroix-A, Lopez C, Lekhal DA, Roques C. Farm Management Practices and Diversity of the Dominant Bacterial Species in Raw Goat’s Milk. Int J Dairy Sci. 2010; 6(1):29-43. doi:10.3923/ijds.2011.29.43.
Barraza A, Montes-Sánchez JJ, Caamal-Chan MG, Loera-Muro A. Characterization of microbial communities from rumen and large intestine of lactating creole goats grazing in arid plant communities. Microbiology. 2021;167(10). doi:10.1099/mic.0.001092.
Neviani E, Bottari B, Lazzi C, Gatti M. New developments in the study of the microbiota of raw-milk, long-ripened cheeses by molecular methods: the case of Grana Padano and Parmigiano Reggiano. Front Microbiol. 2013; 4 (36): 1-14. doi:10.3389/fmicb.2013.00036.
Yang Z, Ni L, Tian W, Chi H. Screening and Identification of Goat-Milk-Derived Lactic Acid Bacteria with Bacteriocin-like Activity and Probiotic Potentials. Microorganisms. 2023-, 11(4):849. doi:10.3390/microorganisms11040849.
Toquet M, Gómez-Martín betaller E. Review of the bacterial composition of healthy milk, mastitis milk and colostrum in small ruminants. Research in Veterinary Science. 2021; 140 1-5 https://doi.org/ 10.1016/j.rvsc.2021.07.022.