Ensayo de PCR en tiempo real basado en SYBR Green para la detección del virus de la diarrea epidémica porcina
Contenido principal del artículo
Resumen
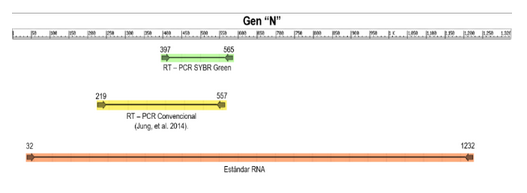
La diarrea epidémica porcina (DEP) es una enfermedad contagiosa producida por el virus del mismo nombre que es clasificado dentro del género Alphacoronavirus, familia Coronaviridae. Este virus produce diarrea, vómitos y pérdida de peso en cerdos de todas las edades, pero en los cerditos neonatos alcanza una morbilidad y mortalidad de hasta el 100%. En Ecuador, fue diagnosticada por primera vez en el 2014 mediante PCR convencional o en punto final. Actualmente existe la necesidad de contar con un ensayo ultrasensible, específico, que permita trabajar un gran número de muestras para facilitar la vigilancia epidemiológica de nuevos brotes y detectar animales portadores. Se desarrolló y evaluó un ensayo de PCR en tiempo real con un colorante intercalador de ADN como el SYBR Green para detectar el virus de la DEP (VDEP) en heces de cerdos, con cebadores que tienen como diana el gen N del virus. En la evaluación de la sensibilidad analítica se utilizó un patrón de ARN del VDEP sintetizado en el laboratorio, determinándose que el límite de detección del ensayo desarrollado fue de 4 copias de ARN del VDEP, comparando con la PCR en punto final que fue 100 veces más sensible, la especificidad del ensayo se basó en la posibilidad de detectar falsos positivos mediante la curva de disociación. Los valores de Ct (valor umbral) en el análisis intra e inter-ensayo mostraron coeficientes de variación válidos. En la evaluación del desempeño diagnóstico, el ensayo detectó 18 muestras positivas de un total de 52 para un 100% de concordancia con el PCR en punto final. Se concluye que el ensayo de PCR en tiempo real con SYBR Green desarrollado es altamente sensible, específico, reproducible y rápido para el diagnóstico del VDEP en homogenados de mucosa intestinal de cerdos.
Detalles del artículo

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Aquellos autores/as que tengan publicaciones con esta revista, aceptan los términos siguientes:
- Los autores/as conservarán sus derechos de autor y garantizarán a la revista el derecho de primera publicación de su obra, el cual estará simultáneamente sujeto a la Licencia Creative Commons Attribution-NonCommercial 4.0 International (CC BY-NC 4.0) que prohíbe el uso comercial de sus publicaciones y permite a terceros compartir la obra siempre que se indique su autor y la primera publicación en esta revista. Bajo esta licencia el autor será libre de:
- Compartir — copiar y redistribuir el material en cualquier medio o formato
- Adaptar — remezclar, transformar y crear a partir del material
- El licenciador no puede revocar estas libertades mientras cumpla con los términos de la licencia
Bajo las siguientes condiciones:
- Reconocimiento — Debe reconocer adecuadamente la autoría, proporcionar un enlace a la licencia e indicar si se han realizado cambios. Puede hacerlo de cualquier manera razonable, pero no de una manera que sugiera que tiene el apoyo del licenciador o lo recibe por el uso que hace.
- NoComercial — No puede utilizar el material para una finalidad comercial.
- No hay restricciones adicionales — No puede aplicar términos legales o medidas tecnológicas que legalmente restrinjan realizar aquello que la licencia permite.
- Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
- Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos telemáticos institucionales o en su página web) antes y durante el proceso de envío, lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).
Citas
Liu Q, Gerdts V. Transmissible gastroenteritis virus of pigs and porcine epidemic diarrhea virus (coronaviridae). Encyclopedia of Virology. Elsevier; 2021. p. 850-3.
Jung K, Saif LJ. Porcine epidemic diarrhea virus infection: Etiology, epidemiology, pathogenesis and immunoprophylaxis. Vet J. 2015;204(2):134-43.
Wang L, Byrum B, Zhang Y. New variant of porcine epidemic diarrhea virus, United States, 2014. Emerging Infect Dis. 2014;20(5):917-9.
Song D, Moon H, Kang B. Porcine epidemic diarrhea: a review of current epidemiology and available vaccines. Clin Exp Vaccine Res. 2015;4(2):166-76.
Garrido A, Barrera M, Vaca M, Acosta A. Primer reporte de diagnóstico molecular de diarrea epidémica porcina en Ecuador. Ecuador es Calidad. 2015;2(2): 15-19.
Barrera M, Garrido-Haro A, Vaca MS, Granda D, Acosta-Batallas A, Pérez LJ. Tracking the origin and deciphering the phylogenetic relationship of porcine epidemic diarrhea virus in Euador. Biomed Res Int. 2017 ;2017:2978718. https://doi.org/10.1155/2017/2978718)
Olech M. Current state of molecular and serological methods for detection of porcine epidemic diarrhea virus. Pathogens. 2022;11(10): 1074 https://doi.org/10.3390/pathogens11101074.
Jung K, Saif LJ, Wang Q. Porcine epidemic diarrhea virus (PEDV): An update on etiology, transmission, pathogenesis, and prevention and control. Virus Res. 2020;286:198045):1-13
Untergasser A, Cutcutache I, Koressaar T, Ye J, Faircloth BC, Remm M, et al. Primer3--new capabilities and interfaces. Nucleic Acids Res. 2012;40(15):e115. doi: 10.1093/nar/gks596. Epub 2012 Jun 22. PMID: 22730293; PMCID: PMC3424584.)
Jung K, Wang Q, Scheuer KA, Lu Z, Zhang Y, Saif LJ. Pathology of US porcine epidemic diarrhea virus strain PC21A in gnotobiotic pigs. Emerging Infect Dis. 2014;20(4):662-5.
Vaerman JL, Saussoy P, Ingargiola I. Evaluation of real-time PCR data. J Biol Regul Homeost Agents. 2004;18(2):212-4.
Vlasova AN, Marthaler D, Wang Q, Culhane MR, Rossow KD, Rovira A, et al. Distinct characteristics and complex evolution of PEDV strains, North America, May 2013-February 2014. Emerging Infect Dis. 2014;20(10):1620-8.
Li F, Zeng Y, Zhang R, Peng K, Jiang C, Xu Z, et al. Genetic variations in S gene of porcine epidemic diarrhoea virus from 2018 in Sichuan Province, China. Vet Med Sci. 2020;6(4):910-8.
Domingo E, Holland JJ. RNA virus mutations and fitness for survival. Annu Rev Microbiol. 1997;51:151-78.
Siddell S, Wege H, ter Meulen V. The structure and replication of coronaviruses. Curr Top Microbiol Immunol. 1982;99:131-63.
Huang Y-W, Dickerman AW, Piñeyro P, Li L, Fang L, Kiehne R, et al. Origin, evolution, and genotyping of emergent porcine epidemic diarrhea virus strains in the United States. MBio. 2013;4(5):e00737-13. doi:10.1128/mBio.00737-13.
Zhou X, Zhang T, Song D, Huang T, Peng Q, Chen Y, et al. Comparison and evaluation of conventional RT-PCR, SYBR green I and TaqMan real-time RT-PCR assays for the detection of porcine epidemic diarrhea virus. Mol Cell Probes. 2017;33:36-41.
Han H-Y, Zheng H-H, Zhao Y, Tian R-B, Xu P-L, Hou H-L, et al. Development of a SYBR green I-based duplex real-time fluorescence quantitative PCR assay for the simultaneous detection of porcine epidemic diarrhea virus and porcine circovirus 3. Mol Cell Probes. 2019;44:44-50.
Zheng L-L, Cui J-T, Han H-Y, Hou H-L, Wang L, Liu F, et al. Development of a duplex SYBR Green? based real-time PCR assay for detection of porcine epidemic diarrhea virus and porcine bocavirus3/4/5. Mol Cell Probes. 2020;51:101544.
Lekanne Deprez RH, Fijnvandraat AC, Ruijter JM, Moorman AFM. Sensitivity and accuracy of quantitative real-time polymerase chain reaction using SYBR green I depend on cDNA synthesis conditions. Anal Biochem. 2002;307(1):63-9.
Ririe KM, Rasmussen RP, Wittwer CT. Product differentiation by analysis of DNA melting curves during the polymerase chain reaction. Anal Biochem. 1997;245(2):154-60.
Duan C. An updated review of porcine deltacoronavirus in terms of prevalence, pathogenicity, pathogenesis and antiviral strategy. Front Vet Sci. 2021;8:811187.
Zhou P, Fan H, Lan T, Yang X-L, Shi W-F, Zhang W, et al. Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin. Nature. 2018 ;556(7700):255-8.
Bolivar AM, Rojas A, Lugo PG. PCR y PCR-Múltiple: parámetros críticos y protocolo de estandarización. Avances en Biomedicina. 20143(1): 25-33
World Organisation for Animal Health. Chapter 2.1.2. Biotechnology Advances in the Diagnosis of Infectious Diseases. Manual of Diagnostic Tests and Vaccines for Terrestrial Animals [Internet]. 2022 [cited 2023 Jun 9]. p. 20. Available from: https://www.woah.org/fileadmin/Home/eng/Health_standards/tahm/2.01.02_BIOTECH_DIAG_INF_DIS.pdf.