Implementación en Cuba de un ensayo molecular para la detección de Brucella spp. en humanos
Contenido principal del artículo
Resumen
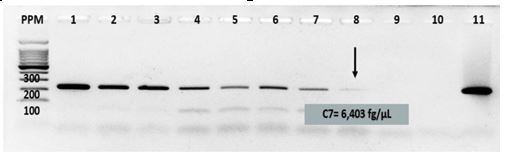
La brucelosis es una zoonosis que puede cursar con síntomas leves o con manifestaciones severas. Las técnicas moleculares son muy sensibles y específicas en la detección oportuna y precoz del ADN de Brucella spp., tanto al inicio de la infección como en las recidivas y en las fallas terapéuticas. El propósito de la presente investigación fue implementar una PCR convencional (bcsp) que permita el diagnóstico oportuno de la brucelosis humana en Cuba, y así contribuir a evitar la aparición de complicaciones que pueden provocar secuelas invalidantes en el paciente. Se realizó una investigación de servicios y sistemas en el Laboratorio Nacional de Referencia de Brucelas del IPK para evaluar la PCR-bcsp. Se determinaron los parámetros de sensibilidad y especificidad analíticas, así como sensibilidad y especificidad diagnósticas. La PCR convencional se aplicó a 86 sueros de casos sospechosos de brucelosis humana que resultaron negativos a los ELISAs IgM e IgG para Brucella . La PCR-bcsp presentó un límite de detección de 6,403 fg/µL, la especificidad analítica fue del 100 %, mientras que la sensibilidad y la especificidad diagnósticas mostraron valores de 96,3 % y 100 %, respectivamente. Tras su aplicación en sueros de casos probables de brucelosis humana negativos por serología se alcanzó un 85 % de positividad. La presente investigación fortaleció el diagnóstico microbiológico de la brucelosis humana en Cuba porque quedó implementada una herramienta molecular que garantiza el diagnóstico temprano de la enfermedad en nuestro medio.
Detalles del artículo

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Aquellos autores/as que tengan publicaciones con esta revista, aceptan los términos siguientes:
- Los autores/as conservarán sus derechos de autor y garantizarán a la revista el derecho de primera publicación de su obra, el cual estará simultáneamente sujeto a la Licencia Creative Commons Attribution-NonCommercial 4.0 International (CC BY-NC 4.0) que prohíbe el uso comercial de sus publicaciones y permite a terceros compartir la obra siempre que se indique su autor y la primera publicación en esta revista. Bajo esta licencia el autor será libre de:
- Compartir — copiar y redistribuir el material en cualquier medio o formato
- Adaptar — remezclar, transformar y crear a partir del material
- El licenciador no puede revocar estas libertades mientras cumpla con los términos de la licencia
Bajo las siguientes condiciones:
- Reconocimiento — Debe reconocer adecuadamente la autoría, proporcionar un enlace a la licencia e indicar si se han realizado cambios. Puede hacerlo de cualquier manera razonable, pero no de una manera que sugiera que tiene el apoyo del licenciador o lo recibe por el uso que hace.
- NoComercial — No puede utilizar el material para una finalidad comercial.
- No hay restricciones adicionales — No puede aplicar términos legales o medidas tecnológicas que legalmente restrinjan realizar aquello que la licencia permite.
- Los autores/as podrán adoptar otros acuerdos de licencia no exclusiva de distribución de la versión de la obra publicada (p. ej.: depositarla en un archivo telemático institucional o publicarla en un volumen monográfico) siempre que se indique la publicación inicial en esta revista.
- Se permite y recomienda a los autores/as difundir su obra a través de Internet (p. ej.: en archivos telemáticos institucionales o en su página web) antes y durante el proceso de envío, lo cual puede producir intercambios interesantes y aumentar las citas de la obra publicada. (Véase El efecto del acceso abierto).
Citas
Laine CG, Johnson VE, Scott HM, ArenasGamboa AM. Global Estimate of Human Brucellosis Incidence. Emerg Infect Dis. 2023; 29(9):1789-97. DOI: 10.3201/eid2909.23 0052. PMID: 37610167; PMCID: PMC1046 1652.
Alharbi MGT, Alanazi AS, Alanazi NF, Alsaleh AK, Alanazl SJ, Alanazi SM, et al. Overview of Brucellosis: Simple Review Article. Pharmacophore. 2022;13(2):101-6. Available in: https://doi.org/10.51847/oSqu90fp0k.
O'Callaghan D. Brucelosis humana: avances recientes y retos futuros.InfectDisPoverty. 2020; 9(101). Available in: https://idpjournal.biomed central.com/articles/10.1186/s40249-020-00715-1
Amjadi O, Rafiei A, Mardani M, Zafari P, Zarifian A. A review of the immunopathogenesis of Brucellosis. Infect Dis. 2019; 51 (5): 321-33. DOI: 10.1080/23744235.2019.1568545
Yagupsky P, Morata P, Colmenero JD. Laboratory Diagnosis of Human Brucellosis. ClinMicrobiol Rev. 2019;33(1):e00073-19. DOI: 10.1128/CMR.00073-19.
Thakura S, Bedia J, Singha R, Gillb K, Arorac A, Kashyap N. Quantitative polymerase chain reaction based quantification of Brucella DNA in serum of pre- and post-therapeutic occupationally exposed infected human population. Journal of Infection and Public Health 2018;11(4): 514-20. DOI:10.1016/j.jiph. 2017.10.004
Projahn M, Hammerl JA, Dieckmann R, Dahouk SA. A Proof of Principle for the Detection of Viable Brucella spp. in Raw Milk by qPCR Targeting Bacteriophages. Microorganisms. 2020;8(9):1326. DOI: 10.3390/microorganisms 8091326.
Wang Y, Wang Z, Zhang Y, Bai L, Zhao Y, Liu C, et al.. Polymerase chain reaction-based assays for the diagnosis of human brucellosis. Ann Clin Microbiol Antimicrob2014; 13:31. DOI: 10.118 6/s12941-014-0031-7
Lugo O, Obregón AM, Echevarría E, Rodríguez Y, Soto Y. Aspectos clínicos y epidemiológicos de la brucelosis humana en tres provincias cubanas (2013-2016). Rev Cubana Med Trop2022; 74(2):e784. Disponible en: https:// revmedtropical.sld.cu/index.php/medtropical/arti cle/view/784.
Baily GG, Krahn JB, Drasar BS, Stoker NG. Detection of Brucella melitensis and Brucella abortus by DNA amplification. J. Trop. Med. Hyg. 1992; 95:271-5. PMID: 1495123.
Mukherjee F, Nagmani K, Surendra KSNL, Subramanian BM, Bahekar VS, Prasad A, et al.. Optimization and validation of a diagnostic realtime PCR for bovine brucellosis. AdvAnim VetSci. 2015; 3(11): 577-87. DOI:10.14737/ journal.aavs/2015/3.11.577.587.
QIAGEN. Manual de instrucciones de uso de QIAsymphony DSP DNA 2015; 40 p. Disponible en: http://www.qiagen.com.
Al- Khater B, Al- Ouqaili M, Al- Anii S. In Term of Molecular Technique, Taxonomic and Diagnostic Aspects of Chronic Human Brucellosis in Ramadi City. Anb Med J. 2020; 12(1):1-12. DOI: 10.33091/AMJ.0101212015.
Talmaci R, Traeger-Synodinos J, Kanavakis E, Coriu D, Colita D, Gavrila L. Scanning of betaglobin gene for identification of beta-thalassemia mutation in Romanian population. J Cell Mol Med. 2004;8(2):232-40. DOI:10.1111/j.1582-49 34.2004.tb00278.x.
Ariza X. Brucelosis. En: Rozman C, Cardellach F, Agustí A, et al., editores. Tratado de Medicina Interna Farreras-Rozman. Vol 2. 19ª ed. España: Elsevier; 2020. p. 2045-7.
Mitka S, Anetakis C, Souliou E, Diza E, Kansouzidou A. Evaluation of different PCR assays for early detection of acute and relapsing brucellosis in humans in comparison with conventional methods. J Clin Microbiol. 2007; 45(4):1211-8. DOI:10.1128/JCM.00010-06
Cecmed. Regulación No. 47-2007: Requisitos para la evaluación del desempeño de los diagnosticadores. Disponibleen: http://www.cecmed.cu/Pages/AmbReg-8.htm.
Zerva L, Bourantas K, Mitka S, Kansouzidou A, Legakis NJ. Serum Is the Preferred Clinical Specimen for Diagnosis of Human Brucellosis by PCR. J. Clin. Microbiol. 2001; 39 (4): 16614. DOI:10.1128/JCM.39.4.1661-1664.2001.
Murray PR, Rosenthal KS, Pfaller MA. Brucella y Francisella. En: Murray PR, Rosenthal KS, Pfaller MA. Microbiologíamédica. 9na ed. España: Elsevier; 2021. p. 383-90.